文章背后的故事:开发国际首个整合宏观/微观进化基因组和功能基因组的综合分析工具PanSyn

作者简介:
于洪伟,遗传学专业2021级博士研究生,师从王师教授和李语丽教授,以第一作者或共同第一作者身份发表SCI论文3篇,获得1项软件著作权,获国家奖学金、山东省研究生优秀成果奖,中国海洋大学优秀研究生等荣誉。
阐释基因组的结构和功能是解码地球生命史和生物多样性的核心。高通量测序技术的革命性突破及各类组学技术的广泛应用,推动生命科学研究快速进入大数据时代。与此同时,基因组共线性分析为比较基因组学研究提供了基本框架。现存物种基因组间/内和祖先与现存物种基因组之间的基因组共线性分析是阐明基因组结构、调控元件及其进化史的关键步骤。在过去的十几年里,利用微观/宏观共线性分析所开展的比较基因组学研究带来了生物学领域诸多重大发现和突破1-6。
2019年夏天,我有幸进入了方宗熙海洋生物进化与发育研究中心王师教授的研究团队(http://www.molevolab.com/),开启了全新的研究生阶段的学术旅程。在基因组学新时代的背景下,王师教授团队经过不断开拓创新取得了一系列具有国际影响力的重要成果,积累了丰富的比较基因组学研究经验。其中,我们团队在2017年利用自开发的宏观共线性分析方法,敏锐地发现虾夷扇贝近乎完美地保留了双侧对称动物祖先的染色体核型。这一发现为研究双侧动物进化提供难得的基因组模型,并为理解动物早期起源和进化机制提供关键线索3(文章背后的故事:The “fossil” genome of scallop Patinopecten yessoensis)。此外,基因组共线性分析也被广泛的应用在我们团队其他重要的研究中并取得关键发现,包括动物血红性状起源演化7,贝类基因组大小适应性进化8以及贝类同形性染色体进化9等。与此同时,基因组宏观共线性分析作为特色分析模块已被纳入我们团队开发的软体动物综合基因组数据库MolluscDB10和与董波教授团队合作开发的进化发育组学数据库EDomics11中,该模块得到了国际同行的肯定和广泛使用。然而,在运用基因组共线性开展相关研究工作的过程中,我们也发现了一些问题。虽然已有许多软件工具可用于基础的基因组共线性分析(例如:MCScanX、DAGchainer、SyMAP等12-14),但它们大多局限于微观层面的分析,即依赖保守的基因顺序信息,使得它们只能用于对亲缘关系密切相关的物种间的比较。然而,理解基因组架构从祖先到现存物种的宏观进化,需要进行深度的系统发育比较,即研究从祖先进化而来的保守的同源基因之间的连锁关系(宏观共线性)。并且越来越多的研究也表明,宏观共线性分析方法在推断动物祖先核型演化历史方面卓有成效2-4,11,15。遗憾的是,随着组装到染色体水平基因组数目的爆炸性增长,目前仍缺乏易于使用且便于个性化定制的宏观共线性分析工具。这成为一个亟待解决的问题。基于我们团队对基因组共线性的强烈兴趣和丰富的研究经验,我们决定着手开发一款用于基因组共线性分析的流程,以填补这一领域的空白。
我最初的想法是开发一个能够有效完成宏观共线性分析的软件。然而,就在我完成了对宏观共线性分析流程的开发并认为该项目即将结束的时候,这个项目的转折点出现了。在与王师教授的日常讨论中,他建议我跳出原有的框架,思考开发一种能够完成全面的基因组共线性分析的综合性工具,而不仅仅是局限于宏观共线性的分析。起初,我不禁怀疑这是不是一个好主意。在这个“被升级”的项目中,我需要思考的首要问题就是:开发一个新的综合性的分析软件真的必要且重要吗?为了回答这个问题,我回顾了基因组共线性概念及应用的发展历史,并调查了50个与其相关的软件工具。我发现,尽管目前已有较多的微观共线性分析工具供我们使用,但这些工具的功能相对单一且有限,难以满足复杂多样的分析需求。最重要的是,目前大多数基因组共线性分析方法主要集中在微观进化尺度上,或仅在微观或宏观单一进化尺度上进行研究,鲜有对两个尺度的综合分析7,8,16,17,严重阻碍了对基因组的泛进化历程及其背后蕴藏的功能意义的全面理解。因此迫切需要开发一种全面且系统的比较基因组学分析工具,以深刻解析基因组结构与功能进化的本质规律。这些研究背景激励着我继续开发和完善PanSyn。
随着基因组共线性分析朝着更加多维化的方向发展,如何打造最全面、最先进的基因组共线性分析工具,已然成为我们面临的一大挑战。万事开头难,我们在框架的初步探索和确定过程中,投入了大量的时间和精力。幸运的是,我们团队长期以来积累的丰富研究经验总能在关键时刻指引我们前行。一个重要的转折点来源于我们团队对扇贝性染色体的研究工作,通过将功能基因组数据与基因组共线性相结合的分析,意外找到了解析扇贝性染色体转换机制的突破口9(文章背后的故事:The ‘forever-young’ secret of scallop sex chromosomes: exceptions to the ‘general’ rules),这暗示我们将功能基因组学数据融入基因组共线性的分析流程,或许能够更好的帮助我们更深入的发掘基因组进化过程中蕴藏的潜在关键决定机制。通过深入的文献调研,我们进一步确认了将多维功能组学数据与基因组共线性进行整合分析的重要性。这一整合分析不仅对于理解基因组共线性存在的功能意义至关重要,更有望助力于深度解析基因功能的调控机制,从而使解密临近基因如何共同调控复杂表型成为可能18。同时,这也使得ENCODE和3D/4D基因组计划中大量增加的组学数据集能够得到有效利用,为生命科学领域的研究提供更为丰富和深入的洞察和见解。基于此,经过与王师教授和李语丽教授的持续且深入的讨论,我们构建了整个流程框架,分为三大核心模块(图1):全面的微观共线性分析模块,先进的宏观共线性分析模块,以及整合微观&宏观共线性及功能组学数据的集成式分析模块。这一框架的搭建,标志着我们向着开发综合性基因组共线性工具的目标迈出了坚实的一步。经过不懈的努力,我们花了大约两年的时间开发了迄今最全面系统的整合宏观/微观进化基因组和功能基因组的综合分析工具(PanSyn,https://github.com/yhw320/PanSyn)。PanSyn不仅包含功能全面的微观共线性分析模块,还极大弥补了宏观共线性分析工具匮乏的局面,并创新提出整合微观和宏观共线性以及调控组学数据的集成式分析模块。回首这段历程,虽然充满了挑战和艰辛,但每一次的突破和收获都让我深感成长。在此,我要特别感谢李语丽教授在文章写作和流程开发等细节方面给予的悉心指导和帮助。
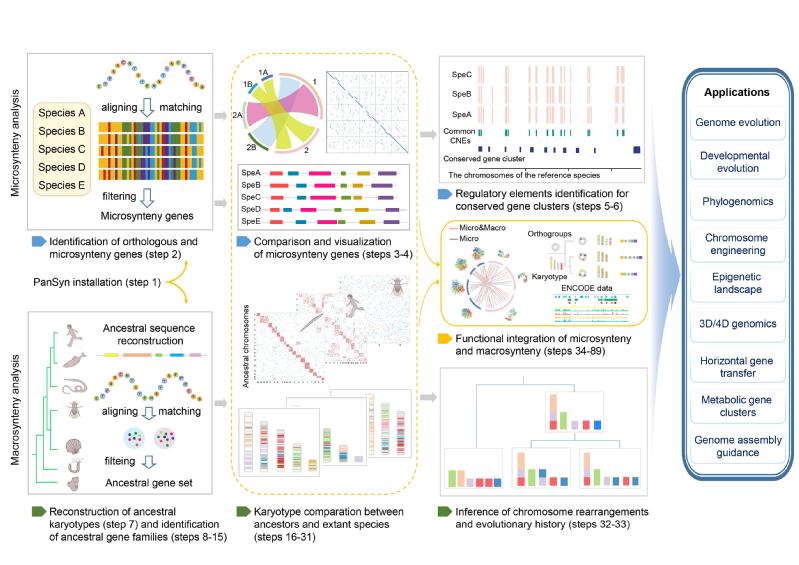
图1. 整合宏观/微观进化基因组和功能基因组的综合分析工具PanSyn流程概览
我们深感欣慰,PanSyn将为全面解析基因组进化和功能、基因资源开发利用等提供有力工具,并热切期盼PanSyn能在生命科学领域催生更多创新发现和重大突破,助力于描绘出整个生命之树的发展演变的全貌。2023年12月中旬,我有幸参加了在新加坡举行的第三届亚太演化生物学大会,并在会上发表了题为“Pan-evolutionary and regulatory genome architecture delineated by integrated macro- and microsynteny approach”的口头报告。在报告结束后,许多与会者纷纷表示对PanSyn的浓厚兴趣和认可,他们的肯定与鼓励是我们未来进行更具影响力科学研究的强大动力。
回顾过去,期间许多徘徊迷茫,幸得王师教授和李语丽教授在整个项目中给予我不断的指导和信心。同时,也要感谢贝类遗传与演化实验室(http://www.molevolab.com)所有小伙伴们的全力支持。此外,我还要向致力于基因组共线性研究的科学家们表示最诚挚的感谢。最后,我衷心感谢众多的国际大型基因组测序项目,赋予了我们攻克重大挑战的机遇和激发创造性灵感的源泉。

图2. 实验室举办Nature Protocols文章的庆祝活动
欢迎大家在相关工作中积极使用和引用PanSyn,如有任何使用问题,请随时与我们联系(hongweiyu@stu.ouc.edu.cn)。
Nature Protocols全文链接
Nature Protocols & Methods Community — Behind the Paper(论文背后的故事):
PanSyn: Elucidation of Pan-evolutionary and Regulatory Genome Architecture
相关支撑论文链接
参考文献
1. Lemons, D. & McGinnis, W. Genomic evolution of Hox gene clusters. Science 313, 1918–1922 (2006).
2. Putnam, N. H. et al. The amphioxus genome and the evolution of the chordate karyotype. Nature 453, 1064–1071 (2008).
3. Wang, S. et al. Scallop genome provides insights into evolution of bilaterian karyotype and development. Nat. Ecol. Evol. 1, 120 (2017).
4. Simakov, O. et al. Deeply conserved synteny resolves early events in vertebrate evolution. Nat. Ecol. Evol. 4, 820–830 (2020).
5. Simion, P. et al. Chromosome-level genome assembly reveals homologous chromosomes and recombination in asexual rotifer Adineta vaga. Sci. Adv. 7, eabg4216 (2021).
6. Nguyen, N. T. T., Vincens, P., Dufayard, J. F., Roest Crollius, H. & Louis, A. Genomicus in 2022: comparative tools for thousands of genomes and reconstructed ancestors. Nucleic Acids Res. 50, D1025–D1031 (2022).
7. Bao, Y. et al. Genomic Insights into the Origin and Evolution of Molluscan Red-Bloodedness in the Blood Clam Tegillarca granosa. Mol Biol Evol. 38, 2351-2365 (2021).
8. Li, Y. et al. Adaptive bird-like genome miniaturization during the evolution of scallop swimming lifestyle. Genomics Proteomics Bioinformatics. 20, 1066–1077 (2022).
9. Han, W. et al. Ancient homomorphy of molluscan sex chromosomes sustained by reversible sex-biased genes and sex determiner translocation. Nat. Ecol. Evol. 6, 1891–1906 (2022).
10. Liu, F. et al. MolluscDB: an integrated functional and evolutionary genomics database for the hyper-diverse animal phylum Mollusca. Nucleic Acids Res. 49(D1), D988-D997 (2021).
11. Wei, J. et al. EDomics: a comprehensive and comparative multi-omics database for animal evo–devo. Nucleic Acids Res. 51, D913–D923 (2023).
12. Wang, Y. et al. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity. Nucleic Acids Res. 40, e49 (2012).
13. Haas, B. J. et al. DAGchainer: a tool for mining segmental genome duplications and synteny. Bioinformatics 20, 3643–3646 (2004).
14. Soderlund, C., Bomhoff, M. & Nelson, W.M. SyMAP v3.4: a turnkey synteny system with application to plant genomes. Nucleic Acids Res. 39, e68 (2011).
15. Simakov, O. et al. Deeply conserved synteny and the evolution of metazoan chromosomes. Sci. Adv. 8, eabi5884 (2022).
16. Xiao, Z. & Lam, H. M. ShinySyn: a Shiny/R application for the interactive visualization and integration of macro- and micro-synteny data. Bioinformatics 38, 4406–4408 (2022).
17. Robert, N. S. M., Sarigol, F., Zieger, E. & Simakov, O. SYNPHONI: scale-free and phylogeny-aware reconstruction of synteny conservation and transformation across animal genomes. Bioinformatics 38, 5434–5436 (2022).
18. Przybyla, L., Gilbert, L.A. A new era in functional genomics screens. Nat Rev Genet. 23, 89–103 (2022).


 English
English 中文
中文

