动植物基因组中由于存在着大量的重复序列,高复杂度等特点,当前仅仅依靠二代测序平台的基因组测序和组装仍然面临许多挑战,许多物种拼接后的基因组存在很多分散的大片段,获得的基因组草图往往难于达到染色体水平。针对这一难题,本研究创建了基于“人工减数分裂”的物理图谱构建新技术-- RadMap,将2b-RAD技术与传统的Happymap实验相结合,可实现对全基因组RAD标签进行精准排序,构建高精度全基因组酶切图谱,用于解决高杂合、高复杂基因组拼接难题。对模式生物拟南芥的验证结果显示,RadMap可显著提升基因组N50等拼接指标达15倍以上,精准度达98.1-98.5%。RadMap对高度片段化的基因组图谱的拼接提升效果优于目前的国际主流的scaffolding技术如BioNano和Hi-C。基于RadMap的组装策略具有实验操作简单、周期短、成本低等优点,能够在不依赖昂贵实验设备条件下实现高效获得高完整性和准确率的参考基因组,在基因组相对复杂并且高度杂合的物种(如海洋无脊椎动物)中有广阔的应用前景。相关研究成果于2017年7月发表于国际遗传学领域权威期刊GENETICS杂志,并被主编选为杂志主页重点推荐论文。
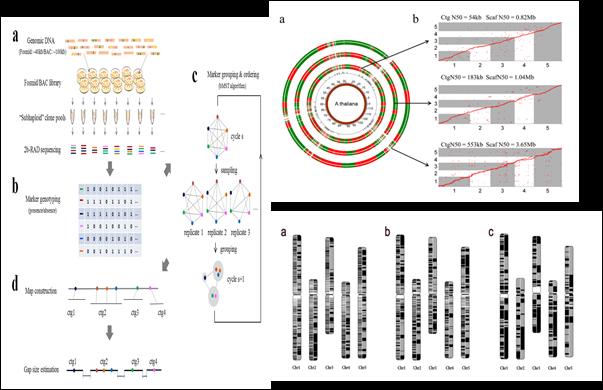
RadMap技术原理(图左)及拟南芥全基因组精细图谱绘制(图右) GENETICS杂志主页重点推荐论文 Dou J#, Dou H#,Mu C, Zhang L, Zhou Z, Chang Y, Wang J, Li T, Li Y, Hu X, Wang S* & Bao Z*.(2017) Whole-genome restriction mapping by “subhaploid”-based RAD sequencing:an efficient and flexible approach for physical mapping and genome scaffolding.Genetics. 206: 1237-1250. 论文链接:http://www.genetics.org/content/early/2017/05/01/genetics.117.200303